Incorporación al IIS-FJD del Dr. Pablo Mínguez como Bioinformático
El Dr Pablo Mínguez, bioinformático, se incorpora al IIS-FJD para participar como investigador en el Proyecto Integrado PIE13/00051 y para desarrollar sus líneas de investigación propias y colaborar en desarrollos del IIS.
Pablo Mínguez es licenciado en Biología (Universidad de Alcalá de Henares) y máster en bioinformática (universidad de East Anglia, Norwich, UK). Realizó su tesis doctoral en el Centro de Investigación Príncipe Felipe (Valencia) en el grupo de Joaquín Dopazo y ha estado los últimos 5 años como postdoc en el EMBL (Heidelberg, Alemania) en el grupo de Peer Bork.
Su línea de investigación e intereses incluyen campos como la genómica y proteómica funcional, el estudio de redes de interacciones entre proteínas y la caracterización funcional de modificaciones post-traduccionales en proteínas, todo ello desde la perspectiva de la Biología de Sistemas y utilizando métodos computacionales, matemáticos y estadísticos. Además tiene experiencia en análisis de varios tipos de experimentos a gran escala y el desarrollo de algoritmos para la descripción de sus resultados.
Ha participado en el desarrollo de diferentes herramientas y bases de datos como la plataforma Babelomics (para el estudio integral de experimentos ómicos), la base de datos STRING (sobre interacciones entre proteínas) o la base de datos PTMcode (sobre asociaciones funcionales entre modificaciones post-traducionales).
Current events
Current events
- 24 de abril de 2024
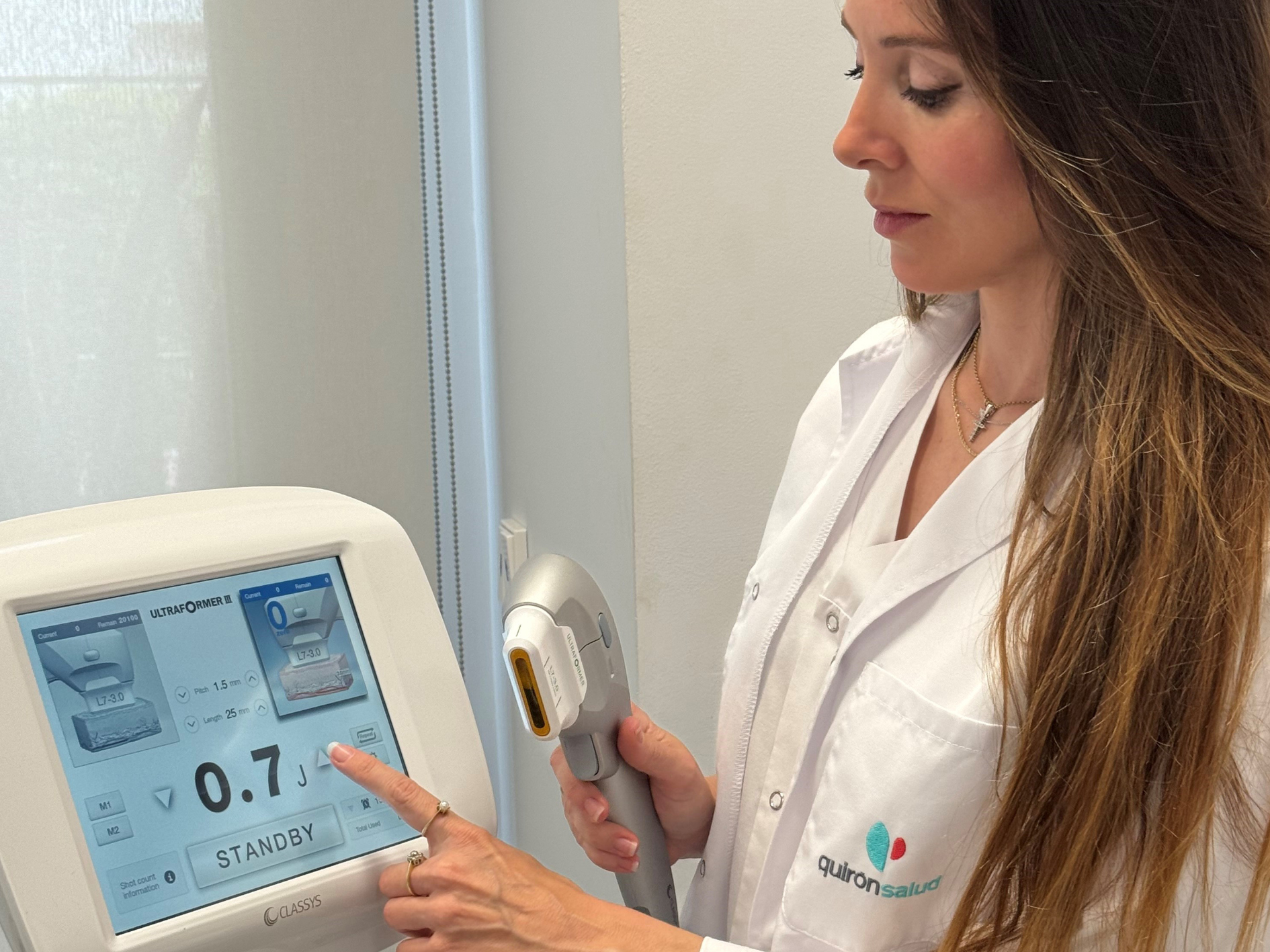
Ultrasonido de alta intensidad, el innovador tratamiento sin cirugía de rejuvenecimiento facial que no...
• Basado en ultrasonidos, no deja signos visibles tras el tratamiento por lo que permite hacer vida normal horas después de la sesión y se puede aplicar en los meses de veran...Hospital Quirónsalud Marbella - 24 de abril de 2024
Quirónsalud, Servicio Médico Oficial del Premier Padel Andalucía Sevilla P2
El torneo, que se disputa del 28 de abril al 5 de mayo, reúne a las principales figuras de este deporte, a nivel nacional e internacional.Quirónsalud - 24 de abril de 2024
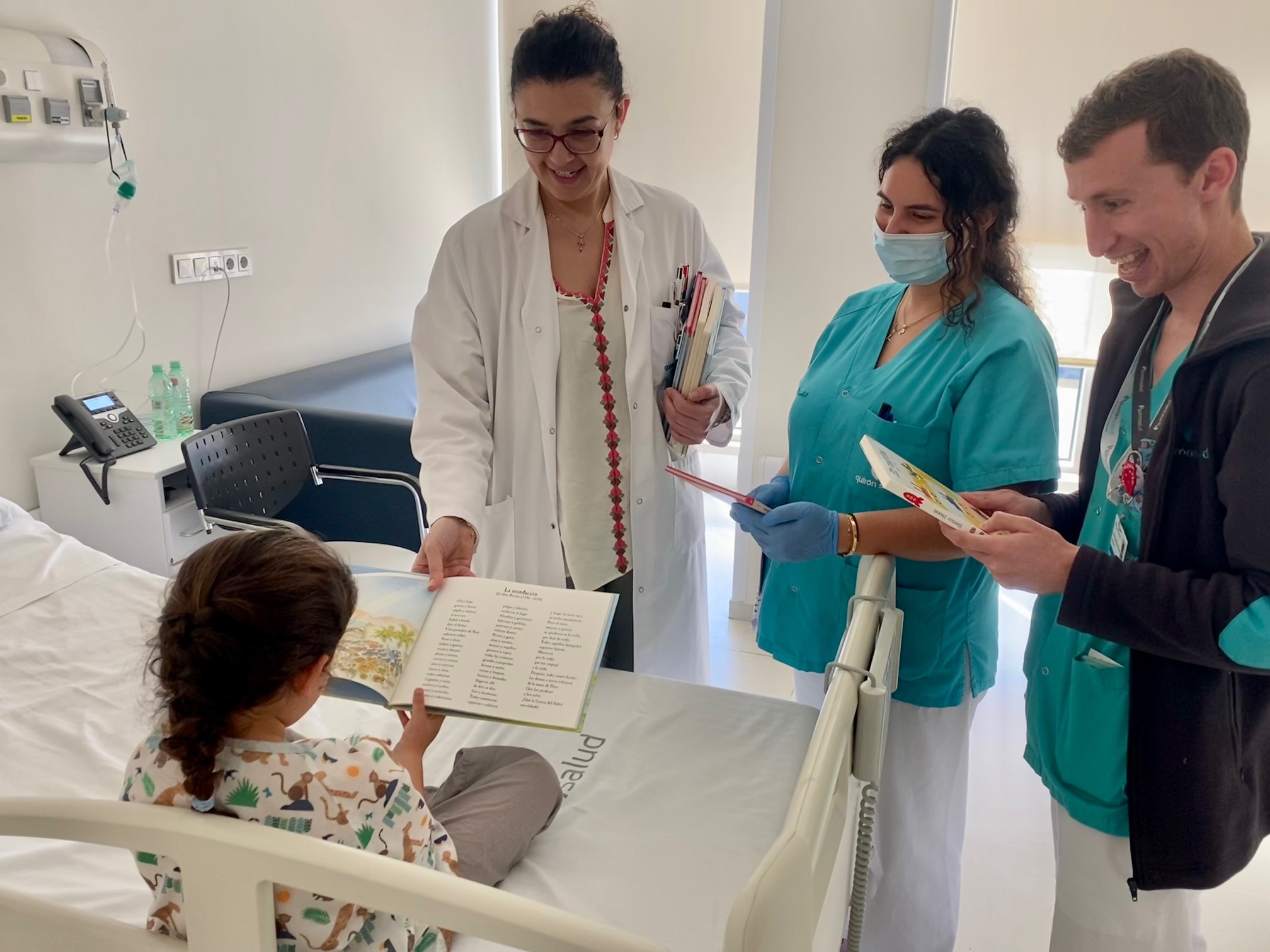
El Hospital Quirónsalud Toledo celebra el Día del Libro con la lectura de ‘Las aventuras de Kenko’
Evadir del ámbito hospitalario, hacer disfrutar con la lectura y fomentar los hábitos saludables, los objetivos de la iniciativa.Hospital Quirónsalud ToledoPediatría y sus Áreas Específicas - 24 de abril de 2024
El Centro Médico Quirónsalud Valdebebas ofrece la posibilidad de conocer la edad metabólica
· Durante los viernes 26 de abril y 10 de mayo en horario de 9.00 a 14.00 horas y los miércoles 8 y 22 de mayo en horario de 15.00 a 20.00 horas. · Las personas interesadas q...Centro Médico Quirónsalud ValdebebasEndocrinología y Nutrición - 23 de abril de 2024
Quirónsalud Infanta Luisa celebra el Día del Libro con sus pacientes hospitalizados para promover los...
Quirónsalud Infanta Luisa regala libros a sus pacientes hospitalizados buscando amenizar así su estancia hospitalariaHospital Quirónsalud Infanta LuisaLogopedia - 24 de abril de 2024
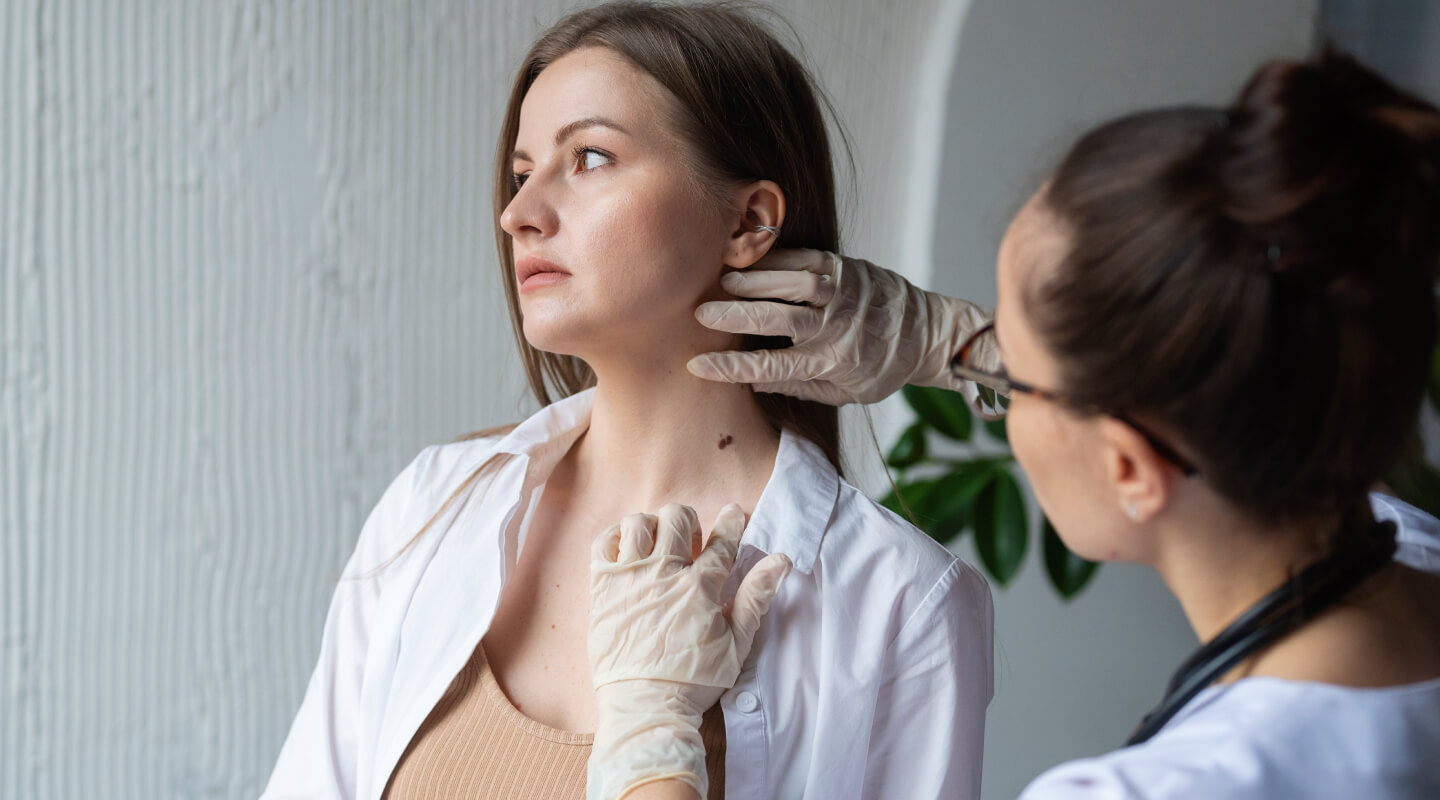
El Hospital Quirónsalud Badalona incorpora un nuevo servicio de Teledermatología para acelerar el diagnóstico...
El nuevo servicio agiliza la atención y respuesta médica, en 48 horas, posibilitando una actuación rápida sobre las lesiones que presenten indicios de malignidad.Hospital Quirónsalud BadalonaDermatología Médico-Quirúrgica y Venereología - 23 de abril de 2024
La Fundación Jiménez Díaz obtiene la acreditación de calidad más reconocida en estudios genéticos, la...
La acreditación, otorgada por la ENAC, refleja la calidad y buenas prácticas del Departamento de Genética del hospitalHospital Universitario Fundación Jiménez Díaz - 22 de abril de 2024
La Fundación Jiménez Díaz obtiene la certificación de calidad más avanzada en sus servicios de UCI Quirúrgica,...
La acreditación es una norma internacional de gestión de la calidad de los cuidados críticosHospital Universitario Fundación Jiménez Díaz - 23 de abril de 2024
Quirónsalud Málaga celebra el Día del Libro con sus pacientes
El personal de Enfermería ha repartido ejemplares cortesía de la Fundación Quirónsalud.Hospital Quirónsalud Málaga - 23 de abril de 2024
Celebramos Sant Jordi con Parada de libros y reparto de rosas
Hoy 23 de abril celebramos Sant Jordi en Quirónsalud Barcelona con el tradicional reparto de rosas y por primera vez con el estreno de una parada de libros tanto para trabaja...Hospital Quirónsalud Barcelona - 23 de abril de 2024
El Hospital Quirónsalud Campo de Gibraltar regala libros a sus pacientes por el Día Internacional del...
El centro de Palmones promueve la lectura entre sus pacientes hospitalizados para tratar de hacer más amena su estancia en el hopspitalHospital Quirónsalud Campo de Gibraltar - 23 de abril de 2024
Quirónsalud Huelva celebra el Día del Libro con sus pacientes hospitalizados
Con motivo del Día Internacional del Libro que se celebra hoy, el Hospital Quirónsalud Huelva ha querido celebrar esta fecha obsequiando a sus pacientes hospitalizados con un...Hospital Quirónsalud Huelva - 23 de abril de 2024
La cirugía con quimioterapia e hipertermia y el uso de dianas moleculares: lo último en oncología
Málaga acoge la II Jornada Nacional Multidisciplinar de Innovación en Cáncer con la participación de más de 100 especialistas en la materiaHospital Quirónsalud MálagaOncología Médica - 23 de abril de 2024
Lectura para celebrar el Día internacional del Libro en el Hospital Quirónsalud Marbella
El objetivo de esta iniciativa es atenuar el estrés de los pacientes y fomentar el hábito de la lectura.Hospital Quirónsalud Marbella - 24 de abril de 2024
El Hospital Universitari General de Catalunya celebra el Sant Jordi con sus pacientes
El 23 de abril, el día de la rosa y el libro, también se ha celebrado en el Hospital Universitari General de Catalunya. Los pacientes ingresados han recibido una rosa junto ...Hospital Universitari General de Catalunya